Et si on voyageait dans le temps avec l’ADN ?
Modifié le :

Article rédigé par CLIO DER SARKISSIAN
Chargée de recherche, CNRSEt si on remontait le temps ? De toutes les machines imaginées par la fiction, il en est une que la science a rendu réelle : la paléogénomique. Cette machine déchiffre l’information génétique encodée dans l’ADN d’organismes morts il y a des centaines, des milliers, voire des millions d’années.
En analysant les molécules d’ADN qui survivent à l’épreuve du temps, les spécialistes sont capables de déchiffrer des génomes complets, accédant ainsi à l’ensemble des séquences d’ADN qui régissent les fonctions biologiques. La paléogénomique est donc un outil très puissant qui permet de suivre avec précision l’évolution du vivant au cours du temps, de retracer nos origines, et même de quantifier notre impact sur la biodiversité. Avec le développement continu des techniques de séquençage d’ADN ancien dans l’ère du big data, la paléogénomique contribue à une connaissance du passé toujours plus riche, offrant des perspectives nouvelles sur les questions que se posent nos sociétés.
L’ADN, molécule du vivant…
L’ADN, ou acide désoxyribonucléique, est une longue molécule, porteuse de l’information génétique chez tous les organismes vivants, bactéries, plantes, animaux. Les techniques classiques dites de « biologie moléculaire » permettent d’isoler et d’analyser les molécules d’ADN à partir de tissus ou de fluides d’individus vivants afin de caractériser les relations génétiques entre les organismes et de comprendre les bases génétiques de l’évolution des populations et des espèces.
… qui survit après la mort
En 1984, l’idée est venue d’appliquer ces mêmes techniques à des restes anciens. Et c’est dans un musée d’histoire naturelle que tout a commencé, celui de Mayence en Allemagne, où le premier spécimen analysé génétiquement fut celui d’un quagga, un membre de la famille des chevaux, ânes et zèbres. Cette espèce, jadis abondante en Afrique du Sud, s’est éteinte en 1883 du fait de sa sur-chasse.
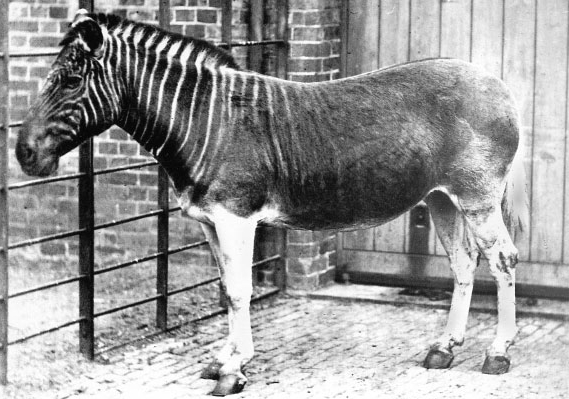
L’étude a montré que des séquences d’ADN proches de celles d’un zèbre actuel pouvaient être retrouvées à partir d’échantillons de muscle prélevés sur un quagga naturalisé il y a 140 ans. Après ce premier tour de chauffe réussi, la communauté scientifique s’est enthousiasmée pour ce nouvel outil qu’elle allait pouvoir appliquer à l’étude des spécimens de zoologie, de paléontologie, d’herbariums, ou de sites archéologiques. Il n’y a rien qui a un jour été vivant qui ne peut en théorie être soumis aux techniques d’ADN ancien, tant que les restes sont préservés jusqu’à nos jours : pour les animaux, os, dents et tartre dentaire, mais aussi peaux, cheveux, poils et plumes, ou encore coquilles d’œuf et coquilles de mollusques, pour les plantes, feuilles, graines, ou bois. Les objets archéologiques comme les bijoux, les outils, les poteries, les parchemins et les tissus anciens sont également des sources d’ADN ancien de par les éléments qui les constituent ou qu’ils ont contenus.

Crédits de gauche à droite : Cavalière sur son cheva – credit : Jacques Sierpinski ; Zèbre de montagne Equus zebra et Rhinocéros noir – Diceros bicornis – crédit : Frédéric Ripoll, Muséum de Toulouse ; Vitrine oiseaux de l’exposition permanente du Muséum de Toulouse – crédit : Frédéric Ripoll, Muséum de Toulouse ; Caméléon, exposition temporaire Savanturiers au Muséum de Toulouse – crédit : Muséum de Toulouse ; Plante herbier credit : Boris Presseq, Muséum de Toulouse ; Volumes de l’herbarium de Picot de Lapeyrousse conservés au Muséum de Toulouse – CC by-sa 4,0 Didier Descouens, via Wikimedia.
Bientôt les limites temporelles de la machine seront testées avec le séquençage de l’ADN de peaux de momies égyptiennes de 2 000 ans ou d’os de dinosaures de 80 millions d’années. L’engouement initial a même ensuite quitté le seul champ de la science pour atteindre le grand public, inspirant le livre à succès Jurassic Park de Michael Crichton en 1990, et son adaptation cinématographique par Steven Spielberg en 1993, deux œuvres de fiction qui imaginent comment les techniques d’ADN ancien peuvent redonner vie à des dinosaures.
Jurassic Park ? Mission Impossible !
Cependant, la communauté scientifique s’est rapidement aperçue que les techniques en vigueur étaient sévèrement limitées. Cela est dû au fait qu’après la mort d’un organisme, son ADN subit des dégradations qui ne sont plus réparées par les mécanismes mis en œuvre par les cellules vivantes. On retrouve alors dans les spécimens anciens des molécules d’ADN très courtes et endommagées chimiquement, rendant le travail expérimental très peu rentable et efficace, et conduisant les spécialistes à commettre de fréquentes erreurs dans leurs tentatives de lecture de l’information que ces molécules anciennes portent. Par conséquent, il n’était alors possible d’analyser que de courtes séquences d’ADN ancien, représentant moins de 0.0005% de la totalité de l’information génétique contenue dans le génome.
Par ailleurs, la dégradation de l’ADN ancien rend les analyses très sensibles à la présence d’ADN moderne, qui peut « contaminer » les expériences. Cette contamination peut intervenir à tout moment après la mort du spécimen et peut provenir du sol du site de fouilles, des produits et matériels utilisés pour analyser le spécimen, ou de tout archéologue, paléontologue ou généticien impliqué dans ces analyses. Les contaminations peuvent être contrôlées et réduites, notamment grâce à l’isolation et au nettoyage intensif des laboratoires où les analyses d’ADN ancien sont réalisées (salle blanche), ou grâce au port d’équipements de protection (gants, masques, combinaison, etc). Avec la mise en lumière du problème de la contamination, de nombreux résultats, dont ceux obtenus à partir d’échantillons de momie ou de dinosaures, ont été contestés car ils n’avaient pas été obtenus dans des conditions conformes aux critères stricts établis pour l’authentification des séquences ADN anciennes. Les problèmes inhérents à l’étude de l’ADN ancien ont longtemps été considérés comme des limites techniques insurmontables à la caractérisation de génomes anciens complets.

© Clio Der Sarkissian

© Clio Der Sarkissian
La révolution du séquençage ADN à haut débit au secours de la paléogénomique
Le développement de technologies de séquençage ADN à haut-débit dans les années 2000 a révolutionné le domaine de l’ADN ancien, permettant, en 2010, la caractérisation du génome du premier humain ancien jamais séquencé, un homme de la culture Saqqaq du Groenland daté de 5 500 ans1. Au cours des dix dernières années, le nombre de génomes anciens séquencés a augmenté de façon exponentielle. Cela a permis de reconstruire les trajectoires évolutives d’un certain nombre d’espèces, dont certaines sont aujourd’hui éteintes, comme les mammouths par exemple. L’ADN ancien a aussi retracé les migrations humaines qui ont conduit au peuplement de la planète. Une autre application de la paléogénomique est l’étude de la modification des espèces animales et végétales au cours des processus de domestication, comme pour le cheval, le chien, le cochon, ou le maïs. Pour finir, l’ADN isolé sur des victimes de pandémies historiques a rendu possible l’identification des bactéries impliquées, entre autres, dans les infections de lèpres et de pestes. Parmi elles, on compte la Peste Noire du XIVe siècle, qui décima près de la moitié de la population de l’Europe, et dont un cimetière a été découvert en 2014 à deux pas du Muséum de Toulouse, rue des Trente-Six Ponts.
La course au génome le plus ancien
Depuis la mise en route de la machine paléogénomique, les spécialistes n’ont eu de cesse de battre un record, celui du génome séquencé le plus ancien. La tâche est loin d’être facile puisqu’il faut trouver un spécimen ayant bénéficié de conditions optimales de conservation lui permettant d’être préservé, avec son ADN, jusqu’à aujourd’hui. Un environnement sec et des températures basses, mais surtout constantes, ralentissent la dégradation post-mortem de l’ADN et augmentent ainsi les chances de battre le record. C’est le cas des sols perpétuellement gelés, appelés pergélisols, dans lesquels ont été mis au jour les restes du cheval vieux de plus de 560 000 ans qui détenait le record du génome le plus ancien depuis 20132. Il a alors été communément admis que même dans des conditions de conservation idéales, les probabilités de retrouver de l’ADN plus vieux qu’un million d’années sont faibles, réduisant à néant les chances de ramener les dinosaures à la vie après 65 millions d’années d’extinction.

© Beth Zaiken / Centre for Palaeogenetics.
Pourtant, en 2022, la barrière du million d’années a bel et bien été franchie avec la publication des génomes de deux mammouths excavés dans le pergélisol sibérien et datés de 1,3 et 1,65 millions d’années. Et en 2023, nouveau record par de l’ADN extrait de sédiments du Groenland. L’âge estimé, 2 millions d’années ! L’analyse de ces données d’un lointain passé nous dépeint une biodiversité surprenante. L’ADN révèle une lignée jusqu’ici inconnue et ancestrale aux premiers mammouths d’Amérique du Nord, et complète ainsi notre connaissance de leur évolution3. Les traces d’ADN laissées dans les sédiments sans qu’aucun fossile ne soit visible à l’œil nu ont permis de reconstruire des écosystèmes passés du Groenland, où vivaient mastodontes, rennes, peupliers, et épicéas depuis longtemps localement disparus4.
Une récompense prestigieuse pour un voyageur du temps
La reconnaissance publique de la paléogénomique en tant que discipline est arrivée en 2022 avec l’attribution du Prix Nobel de Médecine au chercheur suédois Svante Pääbo. Un des premiers mécaniciens de la machine paléogénomique à remonter le temps, il ne s’est pas contenté de son statut de fondateur de la discipline, mais a aussi été l’acteur de la révolution du domaine avec le développement des techniques de séquençage ADN à haut-débit. Le prix couronne le travail de ses équipes qui ont révélé des aspects inédits de l’évolution humaine. Elles ont découvert les Dénisoviens, une population cousine de la nôtre et jusqu’alors jamais identifiée dans le registre fossile. Et si l’on sait aujourd’hui qu’en Europe, chacun de nous sapiens doit environ 2% de son ADN aux Néandertaliens5, une autre population cousine d’Eurasie également éteinte, c’est grâce au travail de ses collègues qui ont mis en évidence les échanges génétiques entre les populations sapiens, dénisovienne et néandertalienne. Le comité Nobel a relevé l’importance pour la santé humaine des implications physiologiques de ces échanges, notamment au niveau des réactions de notre système immunitaire face aux infections, dont celle du COVID.
Le futur d’une science du passé
Les méthodes d’exploration de l’ADN ancien sont constamment améliorées tant par les biologistes moléculaires dans leurs laboratoires que par les spécialistes en bioinformatique devant leurs ordinateurs, ce qui permet de générer et d’analyser de manière plus performante un nombre toujours croissant de données à partir de spécimens de plus en plus vieux. L’interprétation de ces données en collaboration avec des archéologues, morphologistes 3D, et spécialistes en sciences des paléo-environnements promet d’aider à comprendre comment les systèmes biologiques ont répondu aux changements environnementaux et aux activités humaines du passé. Connaître ces processus est particulièrement pertinent pour les sociétés actuelles au vu des changements globaux auxquels nous assistons aujourd’hui avec les effets du réchauffement climatique et de la pollution. Le potentiel de la paléogénomique à retracer le passé souligne la valeur scientifique et l’importance des spécimens conservés dans les Muséums d’Histoire Naturelle comme celui de Toulouse.
Notes de bas de page :
- RASMUSSEN Morten et al., « Ancient human genome sequence of an extinct Palaeo-Eskimo », Nature, n° 463, 2013, p. 757-762. Disponible en ligne, https://doi.org/10.1038/nature08835 [lien valide en octobre 2023].
- ORLANDO Ludovic et al., « Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse », Nature, n° 499, 2013, p. 74-78. Disponible en ligne, https://doi.org/10.1038/nature12323 [lien valide en octobre 2023].
- VAN DER VALK Tom et al., « A Million-year-old DNA sheds light on the genomic history of mammoths », Nature, n° 591, 2023, Article 7849. Disponible en ligne, https://doi.org/10.1038/s41586-021-03224-9 [lien valide en octobre 2023].
- KJÆR Kurt H. et al., « A 2-million-year-old ecosystem in Greenland uncovered by environmental DNA », Nature, n° 612, Article 7939. Disponible en ligne, https://doi.org/10.1038/s41586-022-05453-y [lien valide en octobre 2023].
- GREEN Richard E. et al., « A draft sequence of the Neanderthal Genome », Science, n° 328, 2010, p. 710-722. Disponible en ligne, https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5100745/ [lien valide en octobre 2023].
Pour aller plus loin :
- Livre de Orlando L, 2021, ADN fossile, une machine à remonter le temps. Odile Jacob, p 256
- Émission de radio en podcast. La Terre au carré, par Mathieu Vidard : Quand l’ADN environnemental révèle un écosystème de deux millions d’années
- Invités : Ludovic Orlando et Evelyne Heyer – lundi 9 janvier 2023
- Cours et Séminaire du Collège de France. Lluis Quintana-Murci, chaire Génomique humaine et Evolution. L’histoire de l’humanité vue sous l’angle de la paléogénomique.
- Communiqué de presse de l’Assemblée Nobel à l’occasion de l’attribution du Prix Nobel de Médecine à Svante Pääbo [en anglais]

